A maioria dos medicamentos regularizados e disponíveis para venda Brasil afora levou ao menos dez anos desde o processo de concepção até a chegada às prateleiras das farmácias. É por isso que, em meio a pandemia, uma das estratégias utilizadas pelos cientistas para acelerar esse processo é fazer o rastreamento de remédios já existentes que, embora tenham sido criados para tratar outras doenças, possam ter algum tipo de potencial contra o agente infeccioso em circulação.
O procedimento, conhecido como reposicionamento de fármacos, foi um dos alvos de um estudo norte-americano publicado na revista científica Biomolecules no último dia 23 de maio. A pesquisa in silico (“computacional”) afirma ter identificado 40 compostos derivados de substâncias aprovadas pela Food and Drug Administration (FDA), agência reguladora dos Estados Unidos, e outros 310 sem precedentes entre produtos farmacêuticos regulados e aprovados até o momento que, durante simulações computacionais, demonstraram ter potencial significativo de inibir a atividade do Sars-CoV-2.
Estudos in silico, no entanto, não servem para medir a eficácia ou segurança de um composto. Um de seus objetivos, na verdade, é criar uma espécie de “biblioteca virtual” de substâncias que têm maior probabilidade de se tornarem alvos de medicamentos e poderão ser avaliadas para a realização de testes com animais e humanos no futuro.
Nesse caso, a partir de uma triagem virtual (VS, na sigla em inglês), os pesquisadores construíram uma base de dados com mais de 30 milhões de substâncias com algum nível de potencial contra três proteínas-alvo: a spike (ou “S), usada pelo novo coronavírus para entrar nas células humanas; a serina protease transmembrana II (TMPRSS2), envolvida na fusão entre a membrana do Sars-CoV-2 e a do hospedeiro; e a Mpro, uma enzima catepsina que tem um papel chave no processo de replicação do vírus.
Desse total, 350 apresentaram os melhores resultados em procedimentos de docagem molecular (“docking”, em inglês), uma abordagem que permitiu à equipe modelar qual é o comportamento de cada composto em relação ao trio de proteínas. A maior parte deles (167) foram associados à inibição da acoplagem da proteína S com o receptor ACE2 — presente na superfície de diversas células do corpo —, que corresponde à primeira etapa da infecção pelo novo coronavírus. Outros 40 atuam contra a TMPRSS2 e 103 têm potencial de bloquear a ação da Mpro. Essas três moléculas são consideradas alvos importantes para o desenvolvimento de vacinas e possíveis fármacos contra a Covid-19.
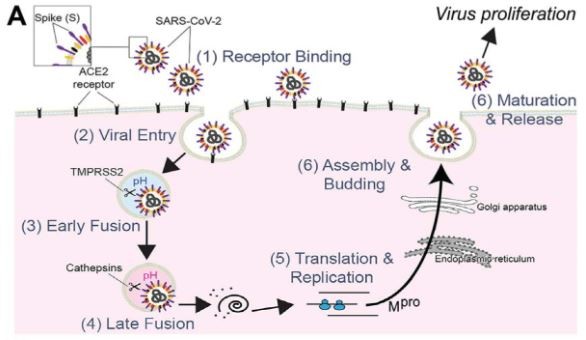
“Se for possível interromper, bloquear ou inibir essas três vias relacionadas às proteínas S, TMPRSS2 e Mpro, então o vírus seria impedido de se proliferar, por isso o que propomos é talvez criar um coquetel, ou seja, diferentes compostos que combinados poderiam ter um efeito sinérgico, em vez de apenas um único isolado”, avalia Thomas Caulfield, professor associado de neurociência na Mayo Clinic e autor sênior da pesquisa, em entrevista a GALILEU.
O pesquisador, no entanto, ressalta que os compostos rastreados não devem ser vistos como parâmetro para uso automático em ensaios clínicos — tampouco consumidos ou prescritos em casos de Covid-19. “Esses resultados são pré-clínicos, os compostos não devem ser candidatos a testes em humanos neste momento. Há um longo caminho até lá”, alerta o bioquímico, que tem vasta experiência em testes in silico e, no estudo, liderou um time de pesquisadores das instituições Mayo Clinic, Universidade da Califórnia em Riverside e Escola de Medicina de Harvard, todas nos Estados Unidos.
Simulações in vitro e mini-fígado 3D
Entre os 350 compostos peneirados pelo estudo por meio de técnicas de Inteligência Artificial (IA), cerca de 11% (40) são derivados de substâncias aprovadas pelo FDA, com a maioria tendo níveis de segurança e tolerabilidade bem estabelecidos por grandes ensaios com humanos (fase III) e análises pós-venda (Fase IV). Os outros 89% (310) são novas entidades químicas (NCEs), isto é, drogas que, até o momento, não têm registros entre produtos farmacêuticos regulados e aprovados.
Para avaliar a toxicidade do segundo grupo, os pesquisadores fizeram experimentos com células humanas infectadas com o Sars-CoV-2 e um pequeno grupo deles (25) também foi selecionado aleatoriamente para ser testado em um micromodelo de fígado humano — um dos principais órgãos responsáveis pela metabolização de medicamentos no corpo.
A minipeça 3D in vitro foi criada em uma superfície do tipo “honeycomb” — menor do que uma ponta de borracha de lápis — por Y. Shrike Zhang, professor assistente na Escola de Medicina de Harvard e bioengenheiro associado do Brigham and Women’s Hospital, que também participou do estudo. “É uma espécie de mimetismo biológico que nos permite imitar um fígado humano, assim podemos aplicar as drogas lá para ver se elas são seguras ou não, e se causam algum efeito deletério”, explica Caulfield.
Segundo a pesquisa, os compostos “não exibiram nenhuma toxicidade significativa” durante essa etapa de triagem pré-clínica, mas testes adicionais deverão ser feitos para esclarecer a hipótese. Os compostos que tiveram melhor desempenho nessa fase serão usados pela equipe no próximo estágio do estudo, que será realizado com camundongos infectados pelo Sars-CoV-2. O modelo animal já está sendo desenvolvido pelos membros do grupo ligados à Universidade da Califórnia em Riverside.
“Todos os 350 compostos são pré-selecionados para serem ‘drug-like’ [termo que se refere às moléculas que contêm características consistentes com a maioria dos medicamentos conhecidos], porque o que fizemos foi comparar as propriedades físicas de medicamentos aprovados pelo FDA com os que selecionamos em nossa abordagem in silico e filtrar aqueles que não tinham semelhanças”, explica Caulfield. “Dessa forma, esperamos ter capturado os compostos com maior probabilidade de serem seguros”.
‘Resultados não se destinam a testes em humanos’
No estudo, os cientistas também destacam que a criação dessa “plataforma de compostos” a partir de triagens virtuais pode ajudar a acelerar as pesquisas em torno de potenciais fármacos contra a Covid-19, enquanto as vacinas ainda não estão disponíveis em larga escala para toda a população.
Segundo Caulfield, os testes in silico tiveram início em março de 2020 e seus primeiros resultados foram obtidos em junho. “A vantagem [dos estudos in silico] é a velocidade. Em três meses, foi possível apresentar um conjunto hipotético de compostos para testes reais”, analisa o cientista.
Embora o método seja capaz de acelerar o processo de descoberta de potenciais fármacos, o pesquisador pondera que as etapas que o sucedem, que costumam levar mais tempo para serem concluídas, não devem ser desrespeitadas. Afinal, somente elas são capazes de atestar a real eficácia e segurança de uma fórmula — e só acontecerão se, antes, esses compostos apresentarem bons resultados nos estágios anteriores.
“Nossos resultados não se destinam a testes em humanos, estamos apenas olhando para novos compostos e drogas existentes do FDA que serão testadas em camundongos infectados para ver se há algum efeito de combinação que possa bloquear a atividade de Covid-19, porque esse é o ponto crucial”, diz Caulfield. “Depois que nosso processo pré-clínico regular for seguido, só então poderemos prosseguir com o processo de ensaio clínico. Portanto, mesmo com a grande necessidade de algo neste momento de pandemia, temos etapas a serem seguidas para garantir a segurança ao público”.
*Com supervisão de Larissa Lopes
Fonte: Revista Galileu











